# Remove warnings
import warnings
warnings.filterwarnings('ignore')Dane ustrukturyzowane
Dane jako zmienne
# variables
customer1_age = 38
customer1_height = 178
customer1_loan = 34.23
customer1_name = 'Zajac'dlaczego do analizy danych nie używamy zmiennych?
Niezależnie od typu analizowanych i przetwarzanych danych w Pythonie możemy zebrać dane i reprezentować je jako pewna formy
listy.
# python lists
customer = [38, 'Divorced', 1, 56.3, ["","",""], {}]
print(customer)# different types in one object
type(customer)dlaczego listy nie są najlepszym miejscem na przechowywanie danych?
Weźmy dwie listy numeryczne
# dwie listy danych
a = [1,2,3]
b = [4,5,6]Typowe operacje na listach w analizach danych
# dodawanie list
print(f"a+b: {a+b}")
# można też użyć metody format
print("a+b: {}".format(a+b))# mnożenie list
try:
print(a*b)
except TypeError:
print("no-defined operation")Każdy obiekt pythonowy można rozszerzyć o nowe metody i atrybuty.
import numpy as np
aa = np.array(a)
bb = np.array(b)
print(aa,bb)print(f"aa+bb: {aa+bb}")
# dodawanie działa
try:
print("="*50)
print(aa*bb)
print("aa*bb - czy to poprawne mnożenie?")
print(np.dot(aa,bb))
print("np.dot - a czy otrzymany wynik też realizuje poprawne mnożenie?")
except TypeError:
print("no-defined operation")
# mnożenie również działa# własności tablic
x = np.array(range(4))
print(x)
x.shapeA = np.array([range(4),range(4)])
# transposition row i -> column j, column j -> row i
A.T# 0-dim object
scalar = np.array(5)
print(f"scalar object dim: {scalar.ndim}")
# 1-dim object
vector_1d = np.array([3, 5, 7])
print(f"vector object dim: {vector_1d.ndim}")
# 2 rows for 3 features
matrix_2d = np.array([[1,2,3],[3,4,5]])
print(f"matrix object dim: {matrix_2d.ndim}")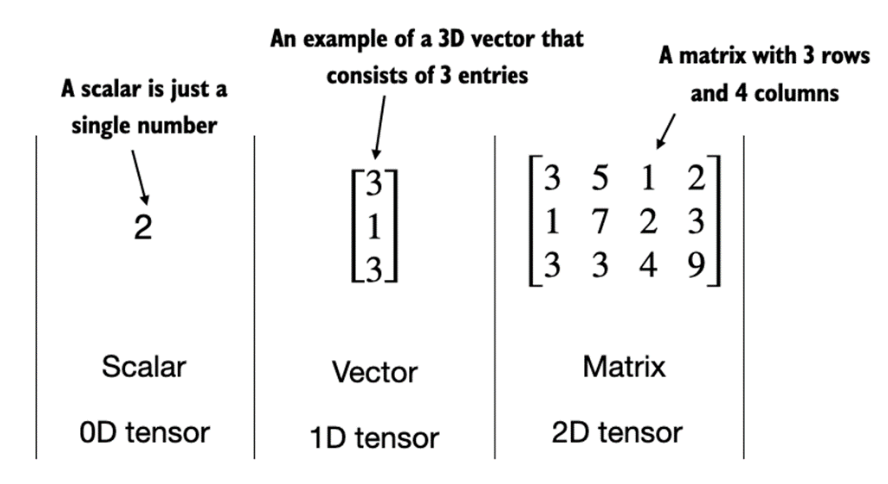
PyTorch
PyTorch is an open-source Python-based deep learning library. PyTorch has been the most widely used deep learning library for research since 2019 by a wide margin. In short, for many practitioners and researchers, PyTorch offers just the right balance between usability and features.
PyTorch is a tensor library that extends the concept of array-oriented programming library NumPy with the additional feature of accelerated computation on GPUs, thus providing a seamless switch between CPUs and GPUs.
PyTorch is an automatic differentiation engine, also known as autograd, which enables the automatic computation of gradients for tensor operations, simplifying backpropagation and model optimization.
PyTorch is a deep learning library, meaning that it offers modular, flexible, and efficient building blocks (including pre-trained models, loss functions, and optimizers) for designing and training a wide range of deep learning models, catering to both researchers and developers.
import torchtorch.cuda.is_available()tensor0d = torch.tensor(1)
tensor1d = torch.tensor([1, 2, 3])
tensor2d = torch.tensor([[1, 2, 2], [3, 4, 5]])
tensor3d = torch.tensor([[[1, 2], [3, 4]], [[5, 6], [7, 8]]])print(tensor1d.dtype)torch.tensor([1.0, 2.0, 3.0]).dtypetensor2dtensor2d.shapeprint(tensor2d.reshape(3, 2))print(tensor2d.T)print(tensor2d.matmul(tensor2d.T))print(tensor2d @ tensor2d.T)szczegółowe info znajdziesz w dokumentacji
Modelowanie danych ustrukturyzowanych
Rozważmy jedną zmienną (xs) od której zależy nasza zmienna wynikowa (ys - target).
xs = np.array([-1,0,1,2,3,4])
ys = np.array([-3,-1,1,3,5,7])Modelem który możemy zastosować jest regresja liniowa.
# Regresja liniowa
import numpy as np
from sklearn.linear_model import LinearRegression
xs = np.array([-1,0,1,2,3,4])
# a raczej
xs = xs.reshape(-1, 1)
ys = np.array([-3, -1, 1, 3, 5, 7])
reg = LinearRegression()
model = reg.fit(xs,ys)
print(f"solution: x1={model.coef_[0]}, x0={reg.intercept_}")
model.predict(np.array([[1],[5]]))Prosty kod realizuje w pełni nasze zadanie znalezienia modelu regresji liniowej.
Do czego może nam posłużyc tak wygenerowany model?
Aby z niego skorzystac potrzebujemy wyeksportować go do pliku.
# save model
import pickle
with open('model.pkl', "wb") as picklefile:
pickle.dump(model, picklefile)Teraz możemy go zaimportować (np na Github) i wykorzystać w innych projektach.
# load model
with open('model.pkl',"rb") as picklefile:
mreg = pickle.load(picklefile)Ale !!! pamiętaj o odtworzeniu środowiska Pythonowego
mreg.predict(xs)siecie neuronowe
from tensorflow.keras import Sequential
from tensorflow.keras.layers import Denseimport tensorflow as tfNa ten problem możemy popatrzeć z innej perspektywy. Sieci neuronowe również potrafią rozwiązywać problemy regresji.
layer_0 = Dense(units=1, input_shape=[1])
model = Sequential([layer_0])
# compilowanie i fitowanie
model.compile(optimizer='sgd', loss='mean_squared_error')
model.fit(xs, ys, epochs=10)print(f"{layer_0.get_weights()}")Inne sposoby pozyskiwania danych
- Gotowe źródła w bibliotekach pythonowych
- Dane z plików zewnętrznych (np. csv, json, txt) z lokalnego dysku lub z internetu
- Dane z bazy danych (np. MySQL, PostgreSQL, MongoDB)
- Dane generowane w sposób sztuczny pod wybrany problem modelowy.
- Strumienie danych
from sklearn.datasets import load_iris
iris = load_iris()# find all keys
iris.keys()# print description
print(iris.DESCR)import pandas as pd
import numpy as np
# create DataFrame
df = pd.DataFrame(data= np.c_[iris['data'], iris['target']],
columns= iris['feature_names'] + ['target'])# show last
df.tail(10)# show info about NaN values and a type of each column.
df.info()# statistics
df.describe()# new features
df['species'] = pd.Categorical.from_codes(iris.target, iris.target_names)# remove features (columns)
df = df.drop(columns=['target'])
# filtering first 100 rows and 4'th columnimport seaborn as sns
import matplotlib.pyplot as plt
sns.set(style="whitegrid", palette="husl")
iris_melt = pd.melt(df, "species", var_name="measurement")
f, ax = plt.subplots(1, figsize=(15,9))
sns.stripplot(x="measurement", y="value", hue="species", data=iris_melt, jitter=True, edgecolor="white", ax=ax)X = df.iloc[:100,[0,2]].values
y = df.iloc[0:100,4].valuesy = np.where(y == 'setosa',-1,1)plt.scatter(X[:50,0],X[:50,1],color='red', marker='o',label='setosa')
plt.scatter(X[50:100,0],X[50:100,1],color='blue', marker='x',label='versicolor')
plt.xlabel('sepal length (cm)')
plt.ylabel('petal length (cm)')
plt.legend(loc='upper left')
plt.show()Dla tego typu danych separowalnych liniowo użyj modelu regresji logistycznej lub sieci neuronowej.
from sklearn.linear_model import Perceptron
per_clf = Perceptron()
per_clf.fit(X,y)
y_pred = per_clf.predict([[2, 0.5],[4,5.5]])
y_predZapis danych i podłączenie do prostej bazy SQL
IRIS_PATH = "https://archive.ics.uci.edu/ml/machine-learning-databases/iris/iris.data"
col_names = ["sepal_length", "sepal_width", "petal_length", "petal_width", "class"]
df = pd.read_csv(IRIS_PATH, names=col_names)# save to sqlite
import sqlite3
# generate database
conn = sqlite3.connect("iris.db")
# pandas to_sql
try:
df.to_sql("iris", conn, index=False)
except:
print("tabela już istnieje")# sql to pandas
result = pd.read_sql("SELECT * FROM iris WHERE sepal_length > 5", conn)result.head(3)# Dane sztucznie generowane
from sklearn import datasets
X, y = datasets.make_classification(n_samples=10**4,
n_features=20, n_informative=2, n_redundant=2)
from sklearn.ensemble import RandomForestClassifier
# podział na zbiór treningowy i testowy
train_samples = 7000 # 70% danych treningowych
X_train = X[:train_samples]
X_test = X[train_samples:]
y_train = y[:train_samples]
y_test = y[train_samples:]
rfc = RandomForestClassifier()
rfc.fit(X_train, y_train)rfc.predict(X_train[0].reshape(1, -1))ZADANIA
- Załaduj plik z danymi
train.csvdo pandasowej ramki danych o nazwie df
## YOUR CODE HERE
df = - Wypisz liczbę wierszy i kolumn załadowanej ramki
## YOUR CODE HERE- Dokonaj czyszczenia braków danych:
- opcja 1 - usuń wiersze zawierające brak danych (
dropna()) - opcja 2 - usuń kolumny zawierające brak danych (
drop()) - opcja 3 - dokonaj imputacji za pomocą wartości średniej (
fillna())
- opcja 1 - usuń wiersze zawierające brak danych (
Które kolumny wybrałeś(aś) do realizacji poszczególnej opcji i dlaczego?
## YOUR CODE HERE- Korzystając z metody
nunique()usuń kolumny, które nie nadają się do modelowania.
## YOUR CODE HERE- Zamień zmienne kategoryjne z wykorzystaniem
LabelEncoderna postać numeryczną
from sklearn.preprocessing import LabelEncoder
le = LabelEncoder()
## YOUR CODE HERE- Wykorzystaj
MinMaxScalerdo transformacji danych zmiennoprzecinkowych do wspólnej skali
from sklearn.preprocessing import MinMaxScaler
## YOUR CODE HERE- Podziel dane na zbiór treningowy (0.8) i testowy (0.2)
from sklearn.model_selection import train_test_split
## YOUR CODE HERE
X_train, X_test, y_train, y_test = train_test_split(...., random_state=44)- Wykorzystując mapowanie możesz dla każdego pasażera przeprowadzić klasyfikację. Funkcja
run()wymaga podania klasyfikatora dla pojedynczego przypadku.- Napisz klasyfikator przypisujący wartość 0 lub 1 w sposób losowy (możesz wykorzystać funkcję
random.randint(0,1)). - Wykonaj fukncję
evaluate()i sprawdź jak dobrze radzi sobie losowy klasyfikator.
- Napisz klasyfikator przypisujący wartość 0 lub 1 w sposób losowy (możesz wykorzystać funkcję
classify = ...def run(f_classify, x):
return list(map(f_classify, x))
def evaluate(predictions, actual):
correct = list(filter(
lambda item: item[0] == item[1],
list(zip(predictions, actual))
))
return f"{len(correct)} poprawnych przewidywan z {len(actual)}. Accuracy ({len(correct)/len(actual)*100:.0f}%)"evaluate(run(classify, X_train.values), y_train.values)